- 病毒包裝服務(wù)
- 慢病毒
- 腺相關(guān)病毒
- 腺病毒
- MMLV逆轉(zhuǎn)錄病毒
- MSCV逆轉(zhuǎn)錄病毒
- 昆蟲桿狀病毒
- 水皰性口炎病毒(VSV)
- 單純皰疹病毒(HSV)
- 痘苗病毒(VACV)
- 重組狂犬病毒(RABV)
- 對照病毒
- 輔助病毒
- 病毒套裝
- 腺相關(guān)病毒
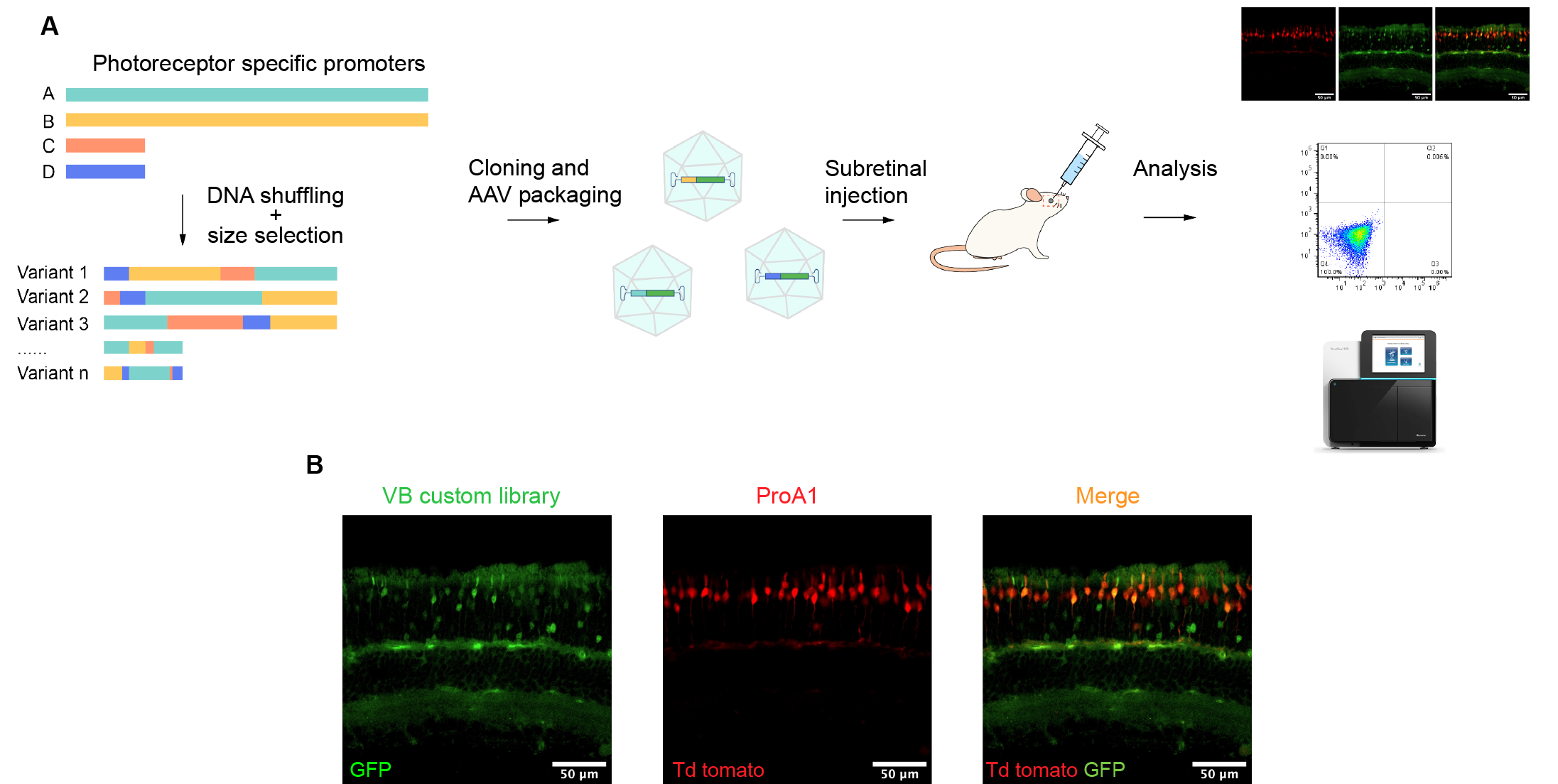
- AAV衣殼定向進(jìn)化
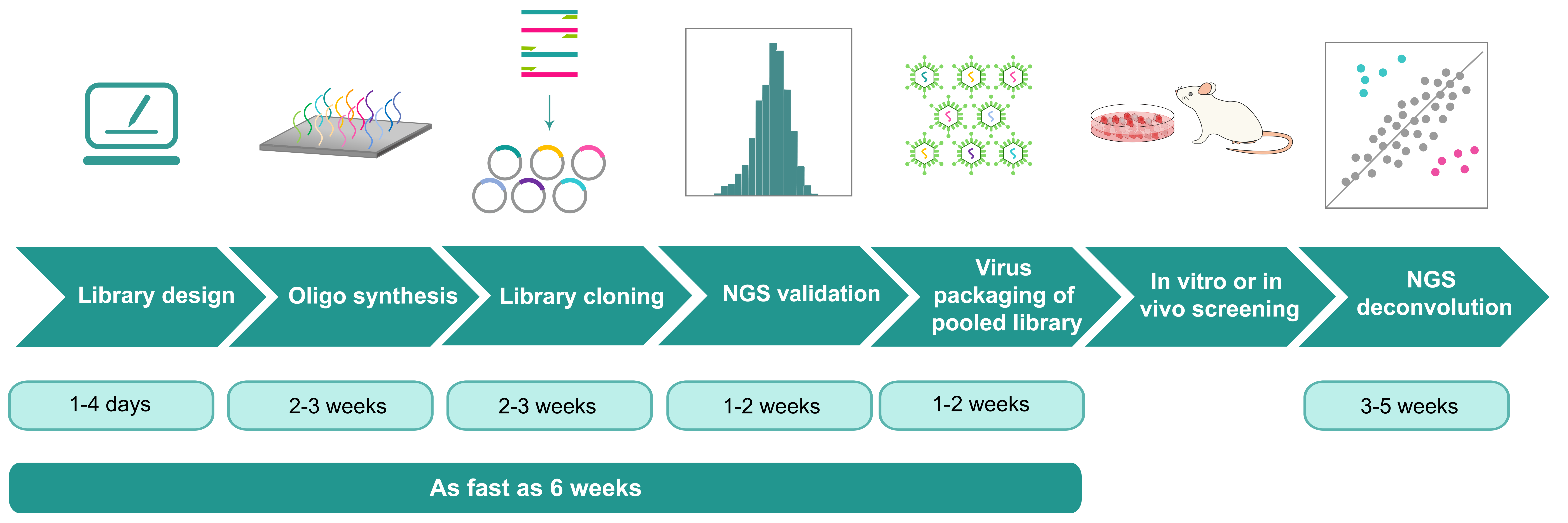
- 文庫篩選
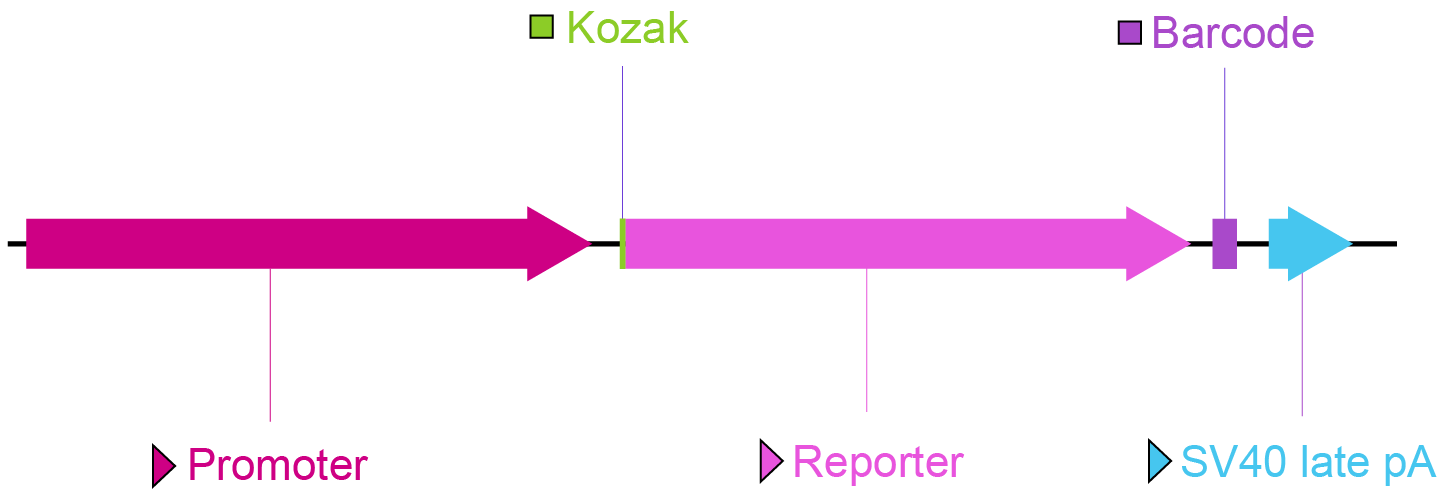
- miniVec®質(zhì)粒載體
- 細(xì)胞穩(wěn)轉(zhuǎn)株構(gòu)建
- shRNA(3+1)套裝
- shRNA基因敲低解決方案
- CRISPR基因編輯解決方案
- 基因誘導(dǎo)表達(dá)解決方案
- COVID-19冠狀病毒解決方案
- 治療性IVT RNA
- AAV衣殼定向進(jìn)化
- 文庫篩選
- AAV組織分布鑒定
- 質(zhì)粒點(diǎn)突變
- 細(xì)胞穩(wěn)轉(zhuǎn)株構(gòu)建
- 重組蛋白表達(dá)
- BAC修飾
- 概況
- cliniVec?
- 工藝開發(fā)
- 分析方法開發(fā)
- 質(zhì)粒制備
- 病毒制備
- IVT mRNA和LNP制備
- GMP設(shè)施
- miniVec®質(zhì)粒載體
- 病毒包裝輔助載體
- AAV血清型彩虹裝
- 特異性啟動子AAV
- AAV類病毒顆粒(VLP)
- 現(xiàn)貨IVT RNA和LNP-mRNA
- 預(yù)設(shè)計(jì)的CRISPR文庫
- 預(yù)設(shè)計(jì)的shRNA文庫
- 對照/輔助載體
- VB UltraStable? 化學(xué)感受態(tài)細(xì)胞
冠狀病毒解決方案